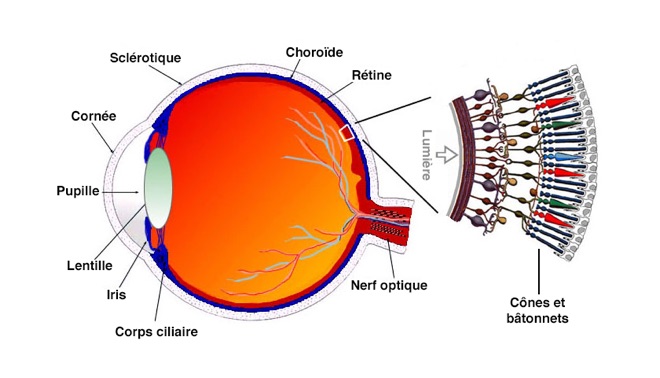
A3: Les opsines, un exemple de famille multigénique.

-
1) Combien y a t-il d’opsines dans l’espèce humaine et quel est leur rôle ?
-
2) Recherchez la définition de famille multigénique et de gènes homologues.
-
3) En vous aidant de la fiche FT Genigen2.pdf , utilisez Génigen2 pour:
-
• Ouvrir dans la banque de séquences le pack famille multigénique Opsines
-
• Aligner les séquences des 3 opsines
-
• Afficher le tableau de comparaison en % d’identité (faites une capture d’écran)
-
• Afficher le phénogramme des 3 Opsines (arbre de parenté génétique)
Ces résultats confirment-ils que les opsines sont issues d’une famille multigénique ?
-
4) En vous aidant de la fiche FT Genigen2.pdf , utilisez Génigen2 pour:
-
• traduire les séquences des 3 opsines (à partir du débutde la séq.)
-
• Aligner les 3 séquences PRO
-
• repérer un groupe de 6 ac. aminés contigus et identiques aux 3 opsines (faites une capture d’écran du secteur concerné et placez les légendes utiles)
-
5) En vous aidant de la fiche FT Genigen2.pdf , utilisez Génigen2 pour:
-
• traduire les séquences des opsines M et L (à partir du débutde la séq.)
-
• Aligner les 2 séquences PRO
-
• repérer un groupe de 4 ac. aminés proches et différents (2+1+1) aux 2 opsines (faites une capture d’écran du secteur concernéet placez les légendes utiles)
-
6) En vous aidant de la fiche Fiche technique Libmol , utilisez LIBMOL pour afficher les 3 opsines :
-
• sur fond noir
-
• en forme RUBAN couleur jaune et le RETINAL en forme «Sphères» avec la couleur captée .
-
• colorez en rose le groupe de 6 ac. aminés communs aux 3 opsines
-
• colorez en rouge le groupe de 4 ac. aminés différents aux opsine M et L
Copiez les images dans le document réponse et apportez les légendes nécessaires.
-
7) En vous aidant de la fiche FT Genigen2.pdf , utilisez Génigen2 pour:
-
• Ouvrir dans la banque de séquences le pack Opsine S chez Primates
-
• Aligner les séquences
-
• Afficher le phénogramme de parenté de l’ Opsine S (arbre de parenté génétique)
Ce résultat est-il en accord avec l’arbre phylogénétique des Primates ci-dessous ?
-
8) L'horloge moléculaire est une hypothèse selon laquelle les mutations génétiques s'accumulent dans un génome à une vitesse constante. Elle permet ainsi théoriquement, en reliant le taux de mutation des gènes à la différence génétique entre espèces proches, d'établir une échelle chronologique approximative de la divergence de ces espèces.
-
•Sachant qu’on estime le taux de mutation dans les gènes de l’opsine à 0,05% par Millions d’années ,
-
•Alignez les séquences des gène M et L puis affichez la dissemblance
-
•A partir du résultat, calculez la date de séparation des gènes M/L
-
•Ce résultat est-il en accord avec les données sur les opsines des Primates et les découvertes de 2010 et les découvertes récentes de paléontologie ?
Les opsines sont une famille de protéines photosensibles que l’on trouve dans la rétine de la plupart des animaux.